プロトコール
こちらでは、DNA修復の基本プロトコールを掲載しています。
近年、色々な研究分野でDNA損傷や修復が関わることが分かってきており、DNA修復分野以外の研究者も、DNA修復のアッセイをすることが多くなっています。ここではこれからDNA修復アッセイを始める人向けに、プロトコールと注意点を記載していますので、ぜひご参照ください。これらのプロトコールや注意点を参照して論文を執筆される際は、関連する文献を引用していただけたら幸いです。
・γH2AXアッセイ
DNA二本鎖切断(DSB)を検出する場合、γH2AXアッセイが最も有名でよく用いられます。
プロトコールはこちら
カバースリップの置き方の動画
抗体wash中に細胞が流れて失いやすい場合のwashの動画
参考文献 Lobrich et al, PMID: 20139725, Shibata et al, PMID: 21317870
*注意すべき点(必見!)
1.γH2AXがfoci(粒々)ではない場合は、DSBではない可能性があります。例えばUVで大量のSSBや塩基損傷が生じた場合は核全体がγH2AXで明るく光ります。S期のDNA複製の時も核全体がγH2AXで明るく光ります。DNA損傷とは関係なく、クロマチン構造が変化したときもγH2AXが検出されると言われています。γH2AXがfociではなく核全体で光っている場合は、DSBではない可能性が高いので下記の方法で確認しましょう。
解決法1)低doseで検証。DSB量が多すぎるかもしれないので、X線なら2-3 Gyの低線量で試しましょう。
解決法2)53BP1 fociを検出しましょう。γH2AX foci = 53BP1 fociとなり、それぞれのfociは同じ場所にあり、1対1の関係になります。核全体がγH2AXで明るく染まり、53BP1は数個のfociだとしたら、53BP1 fociのみにDSBがあると言えます。
2.細胞周期が異なると、当然DNA量が異なるので、γH2AX foci数は変わります。G1とG2を比較した場合、G2細胞はG1細胞の2倍のDNA量があります。つまり、G2細胞はG1細胞の2倍のγH2AX fociが観察されます。例えばX線照射後、コントロール細胞ではG1期で細胞周期が止まり、対象とする〇〇遺伝子ノックダウン細胞ではG1期チェックポイントが効かずにS/G2期に移行してG2期で止まる場合があります。X線照射後24時間の時点で、コントロール細胞はほとんどG1期、〇〇遺伝子ノックダウン細胞ではほとんどG2期、となっていると、〇〇遺伝子ノックダウン細胞ではγH2AX foci数が2倍に見える事になります。これは修復欠損で起こるわけではなく、細胞周期チェックポイントの欠損で起こるということになります。γH2AX fociにて解析を行う場合は、常に細胞周期マーカーと合わせて解析することを強く薦めます。
解決法1)γH2AX、CENPF(G2期マーカー)、EdU(S期マーカー)と染色しましょう。
参考文献 Lobrich et al, PMID: 20139725, Yasuhara et al, PMID: 30245011
・微小核(micronculei)アッセイ
DNA損傷後のある一定時間(24 h, 48 h, 72 h)で固定し、DAPI染色により検出します。微小核は細胞分裂の際に生じます。そのため、DNA損傷後のG2/Mチェックポイント停止が解除されて細胞分裂が起こった後でなければ発生しません。このような理由から、DNA損傷後の24 h, 48 h, 72 hなど遅い時間帯で検出します。40xレンズなどで50-100細胞が視野に入る条件でDAPIを撮影し、PC画面上で、それぞれ細胞1個が有する微小核の数をカウントします。カウントした細胞にはphotoshopなどで赤印をつけておくとカウントしやすいでしょう。一般的には200細胞以上をカウントします。例えば4つの視野の画像をカウントし、4つ目の画像で200細胞を超えたらその画像でカウントを終了します。この場合、カウントした細胞数は例えば231細胞などになります。
*注意すべき点(必見!)
いくつかの論文で、微小核ではなく、アポトーシスの核を数えている論文を見かけます。アポトーシス由来の核断片はクロマチン凝集が起きているため非常に明るいDAPIシグナルになっています。実際の微小核の画像とアポトーシスの画像は下記の論文を参照してください。全く性質の異なるものを解析してしまうので、結果の解釈が大きく変わってしまいます。
微小核の参考文献 Shibata et al, PMID: 31513320
DAPIでの検出やアポトーシスの参考文献 PMID: 29155723
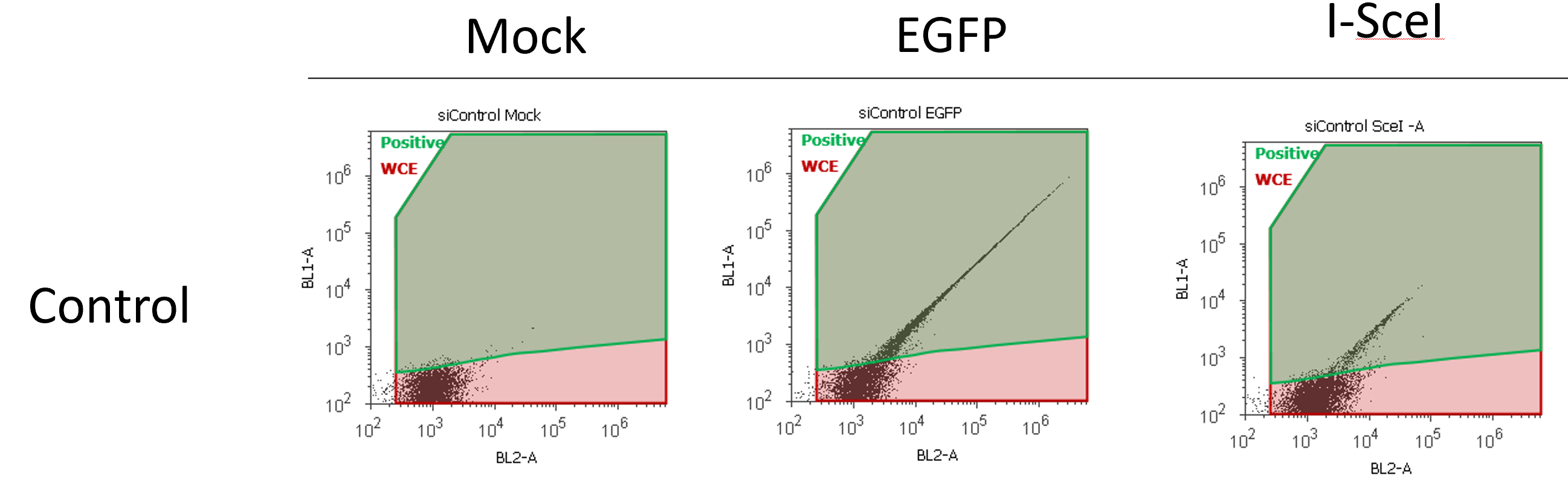
・DR-GFPアッセイ
HR(相同組換え)の頻度を測定するためにMaria Jasinらによって開発された方法です。実験系がうまくいっているか確認するため、BRCA1、BRCA2、RAD51、CtIPなどをノックダウンした細胞を必ずポジティブコントロールとして行いましょう。
用意するサンプルは、
1. Mock(transfection試薬のみ)
2. EGFP transfection
3. I-SceI transfection
フローサイトメトリー後、Mockの細胞群をEGFP(-)としthresholdを設定し、EGFP(+) をゲートします。I-SceI後のEGFPシグナルは弱いですが、Mockで設定したthresholdより上であれば陽性とします。

*注意すべき点(必見!)
HRはS/G2期でのみしか起きません。そのため○○遺伝子のノックダウンにより細胞周期が停止する場合はこの実験系は使えません。以前に転写因子のノックダウンでHR効率が下がっているように見えた時がありましたが、単純に細胞周期が停止し、G1期細胞にだけなった結果だということがありました。フローサイトメトリーなどで細胞周期が変化していないことを併せて確認することが重要です。
EGFP単独のサンプルも必ず用意してください。アウトプットがCMVプロモーターを使ったEGFPの発現細胞になっているため、ノックダウンした遺伝子が転写にも関わる場合だと、EGFP発現量が下がったり上がったりします。その補正が必要ですので、EGFP単独のサンプルでノーマライズする必要があります。
同様なGFPなどの蛍光を使ったNHEJ, Alt-EJ, SSAを検出するレポーターアッセイがありますので、そちらも上記と同様、実験系を理解した上で用いましょう。NHEJのためのポジティブコントロールには、DNA-PK阻害剤や、XLFまたはXRCC4のノックダウンが良いでしょう。一方、Ku80のノックダウンによってNHEJ効率は下がったように見えますが、DSB末端のresectionが増えてGFPが光らなくなるだけで、γH2AX fociで確認すると連結効率は変わっていない場合があります。NHEJの場合はG1期細胞、HRの場合はG2期細胞のγH2AX fociアッセイの結果と合わせて検討することが重要です。
・DSB end resectionを見るためのRPA or RAD51アッセイ
準備中